空間轉(zhuǎn)錄組數(shù)據(jù)分析的核心是根據(jù)每個芯片上每個 spot 的基因表達(dá)信息進(jìn)行聚類,然后將 spot 根據(jù)坐標(biāo)位置序列放回到組織的圖像上,同時可以對每個 gene 在組織上表達(dá)的空間位置進(jìn)行定位。
獲得測序數(shù)據(jù)后,首先利用 Space?Ranger? 軟件可以自動化的對圖像進(jìn)行處理、數(shù)據(jù)比對和 Barcode 處理。另外一個軟件 Loupe?Cell?Browser 是一個適用于 Windows 和 MacOS 的桌面應(yīng)用程序,它可以快速、輕松地可視化和分析 10X?Visium 數(shù)據(jù)。伯豪生物除了提供 spot 基因數(shù)和 UMI 數(shù)統(tǒng)計(jì)、切片 spot 聚類和聚類亞群 marker 基因分析等基礎(chǔ)和高級分外,同時還提供個性化分析,如特定 pathway 功能富集分析等。
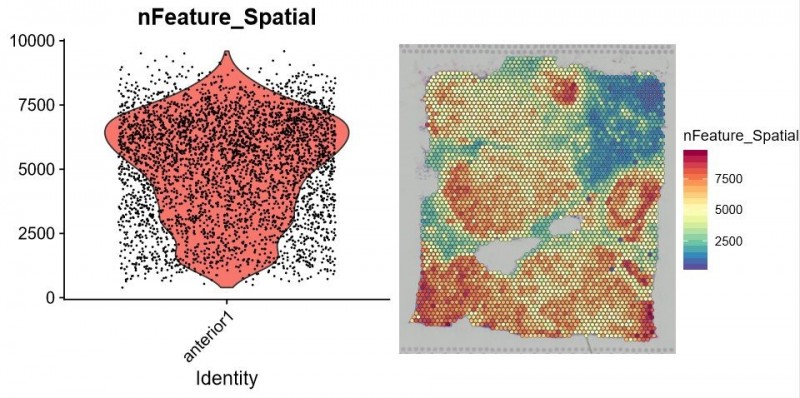
圖 ? 每個 spot 特異表達(dá)的基因數(shù)統(tǒng)計(jì)
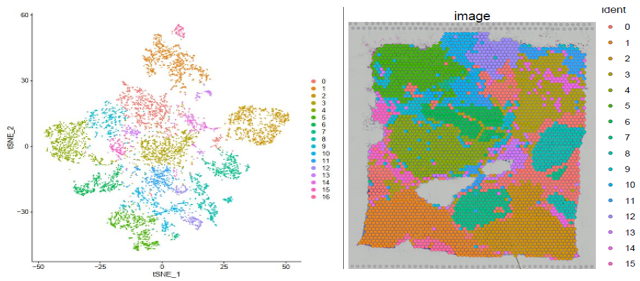
圖 ? 聚類結(jié)果及切片 spot 位置分布展示

圖 特定 pathway 功能富集分析
結(jié)合組織區(qū)域分布對數(shù)據(jù)進(jìn)行挖掘
大部分組織其實(shí)是有其特定的區(qū)域劃分的,比如說大腦里有皮層、丘腦、海馬、脈絡(luò)叢等多個區(qū)域。將組織的區(qū)域劃分和亞群(或細(xì)胞類型)的分布結(jié)合起來還是能發(fā)現(xiàn)很多有價(jià)值的信息的。
可以根據(jù)不同區(qū)域特異表達(dá)的 maker 基因的分布來判斷每個區(qū)域在組織切片上的位置。例如皮層 marker 基因 STX1A 的表達(dá)分布,海馬 marker 基因 HPCA 的表達(dá)分布等
結(jié)合病理學(xué)特征對數(shù)據(jù)進(jìn)行挖掘
空間轉(zhuǎn)錄組技術(shù)正真的精髓不是研究細(xì)胞亞群的分布,而在于將它在空間位置上體現(xiàn)的異質(zhì)性跟組織病理學(xué)特征的分布進(jìn)行結(jié)合,挖掘在不同病理學(xué)特征下轉(zhuǎn)錄組學(xué)的差異。這對于研究疾病病變的機(jī)制、幫助臨床實(shí)現(xiàn)更好的患者分子分型、以及空間位置 Biomarker 的挖掘方面都是非常有價(jià)值的。通過手動把這些區(qū)域圈出來進(jìn)行轉(zhuǎn)錄組層面的比較,找出不同病灶區(qū)的特異性 marker,分析疾病在一步步發(fā)展進(jìn)程中生物學(xué)功能的變化,甚至可以思考一下是否能找出一些關(guān)鍵性因子來阻斷疾病的進(jìn)展。
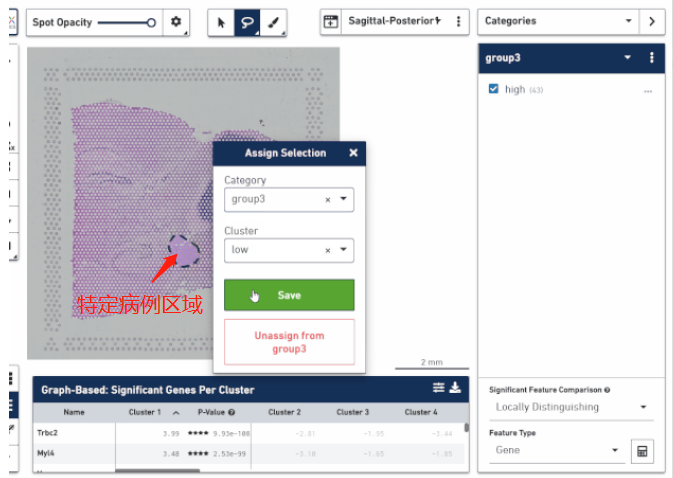
圖 ? 根據(jù)病理信息選取特定區(qū)域分析
空間轉(zhuǎn)錄組聯(lián)合單細(xì)胞 RNA 測序解析細(xì)胞類型的空間位置信息(Multimodal?intersection?analysis,MIA)
空間轉(zhuǎn)錄組測序可以獲得不同基因在組織切片上的空間位置信息,但不能獲得詳細(xì)的細(xì)胞類群信息(空間轉(zhuǎn)錄組不是單細(xì)胞分辨率,只能粗略的分析切片上不同位置的細(xì)胞類型)。因此,需要借助但細(xì)胞測序數(shù)據(jù)來分析細(xì)胞類型,然后通過生物信息學(xué)的分析方法將單細(xì)胞類群映射到空間轉(zhuǎn)錄組數(shù)據(jù)上。
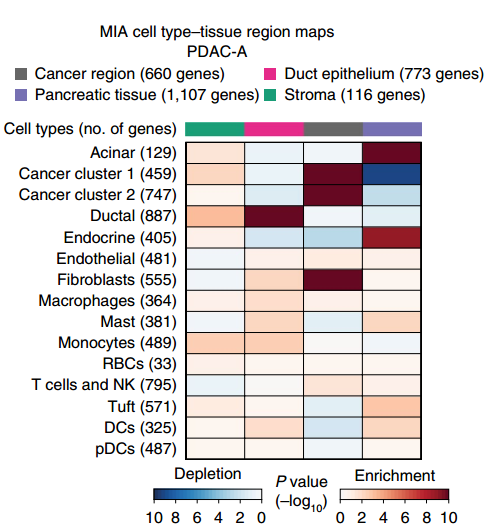
圖 ?MIA 熱圖
備注:MIA 熱圖,上方的顏色條反映了 ST 區(qū)域的子聚類(cancer?region,Pancreatic?tissue,Duct?epithelium 和 stroma)。左側(cè)代表不同的細(xì)胞類群。色塊代表 enrichment 或者 depletion。Enrichment 代表該細(xì)胞類群富集到了該區(qū)域。Depletion 代表該細(xì)胞類群在該區(qū)域缺失。
更多伯豪生物人工服務(wù):


